Festlegen von Achsengrenzen in ggplot2-R-Diagrammen
-
Grenzen der x-Achse mit
scale_x_continuousinggplot2setzen -
Verwenden Sie
coord_cartesian, um beide Achsen inggplot2zu begrenzen
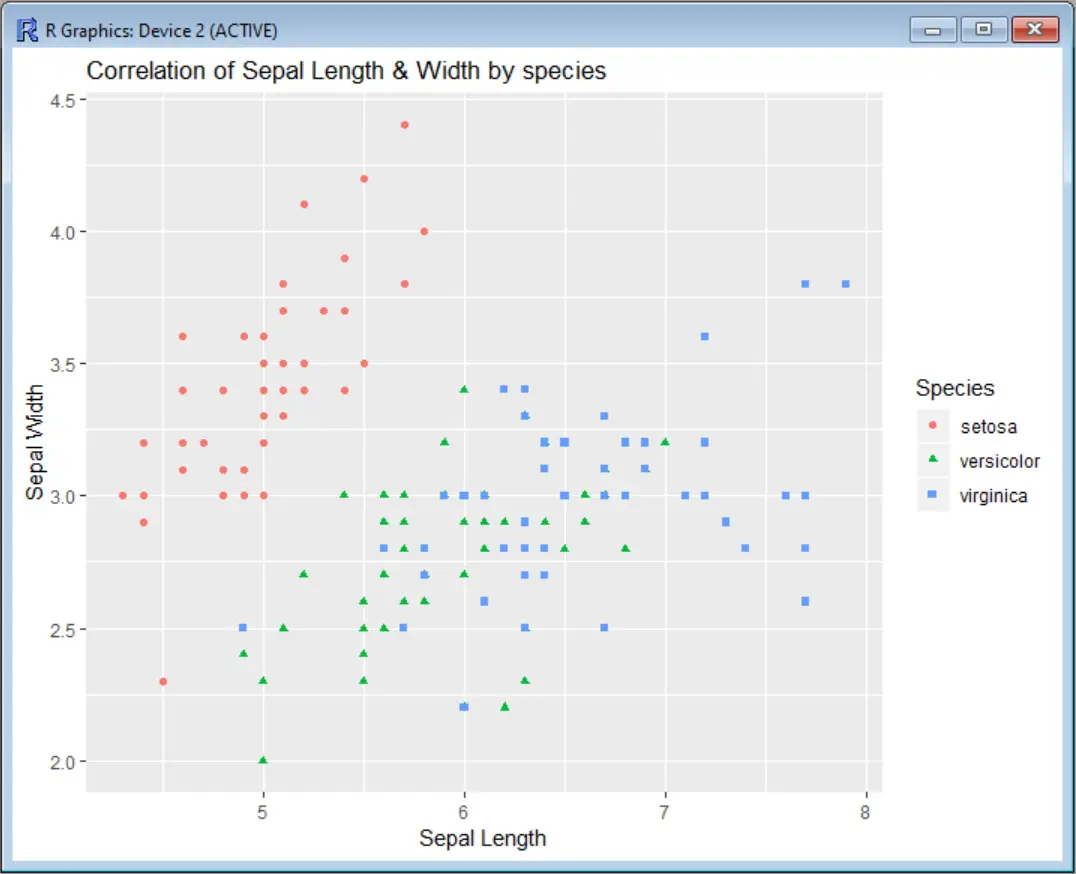
Schauen wir uns zunächst unseren Scatter Plot an, ohne eine Achsenbegrenzung zu setzen. In diesem Beispiel verwenden wir den Iris-Datensatz, um die Werte für die Kelchblattbreite und die Kelchblattbreite zu korrelieren, wobei wir die Arten nach Farbe kategorisieren. Bevor wir die Funktion ggplot aufrufen, müssen wir das Paket ggplot2 installieren und die entsprechende Bibliothek laden:
install.packages("ggplot2")
library(ggplot2)
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species")
Ausgabe:

Nehmen wir nun an, wir wollen die x-Achse (Sepal-Länge) begrenzen, so dass der Plot nur den Bereich zwischen x=5 und x=7 zeigt. Wir haben zwei Möglichkeiten, dies zu tun: mit scale_x_continuous oder mit coord_cartesian.
Bei scale_x_continuous werden alle Datenpunkte entfernt, die außerhalb des angegebenen Bereichs für die gegebene Achse liegen, während bei coord_cartesian nur der sichtbare Bereich angepasst wird. In den meisten Fällen werden beide Optionen die gleichen Ergebnisse liefern. Wenn Sie aber zufällig etwas an die Daten anpassen, würden sich die angepassten Werte wahrscheinlich ändern.
Grenzen der x-Achse mit scale_x_continuous in ggplot2 setzen
Um scale_x_continuous zu verwenden, müssen Sie einen Vektor mit den unteren und oberen Grenzen der x-Achse angeben, etwa so:
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
scale_x_continuous(limits = c(5, 7))
Ausgabe:

Sie erhalten auch eine Warnmeldung, die Ihnen mitteilt, wie viele Datenpunkte aus dem Diagramm entfernt werden:
Warning message:
Removed 34 rows containing missing values (geom_point).
Falls Sie auch die y-Achse begrenzen wollen, können Sie auf die gleiche Weise scale_y_continuous hinzufügen, wie in diesem Beispiel:
scatter-<--ggplot(data=iris,-aes(x-=-Sepal.Length,-y-=-Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
scale_x_continuous(limits = c(5, 7)) +
scale_y_continuous(limits = c(2.5, 4))
Die Ausgabe zeigt dann die Begrenzungen in beiden Achsen an:

Sie können auch die Shorthand-Funktionen xlim und ylim anstelle von scale_x_continuous und scale_y_continuous verwenden, um den Befehl etwas kürzer zu machen:
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
xlim(5, 7) + ylim(2.5, 4)
Verwenden Sie coord_cartesian, um beide Achsen in ggplot2 zu begrenzen
Wenn Sie coord_cartesian anstelle von scale_x_continuous und scale_y_continuous verwenden wollen, würde der Befehl wie folgt aussehen:
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
coord_cartesian(xlim = c(5, 7), ylim = c(2.5, 4))
Die Plot-Ausgabe ist dann die gleiche.
Im RStudio Cheatsheet von ggplot2 finden Sie eine visuelle Erläuterung für die Verwendung aller Optionen für diesen Befehl.